近日,我校计算机科学与工程学院彭理副教授团队在智能生物信息处理领域取得新进展,相关研究成果论文“Predicting CircRNA-Disease associations via feature convolution learning with heterogeneous graph attention network” 发表在国际顶级期刊《IEEE Journal of Biomedical and Health Informatics》上,该期刊属于中科院一区,Top期刊,影响因子:7.021。
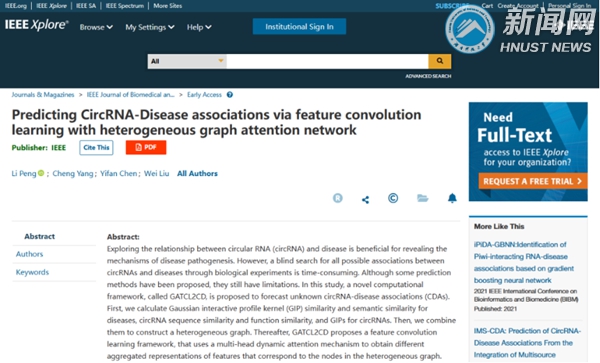
▲学术论文期刊展示

▲期刊分区
在这项研究中,提出了一种新的环状RNA-疾病关联计算框架,称为GATCL2CD,该算法基于异构图注意力网络的特征卷积学习模型对未知的环状RNA -疾病关联进行预测。该方法首先计算了环状RNA序列、功能、高斯交互谱核相似性,以及疾病语义、高斯交互谱核相似性,在此基础之上利用相似性阈值划分获得关联子图,进而构建异构图。在异构图中利用多头动态注意力机制获得邻居节点特征的不同聚合表示,利用具有不同大小卷积核的单层卷积网络从堆叠的特征聚合表示中提取节点的高阶特征信息,然后,利用高阶特征信息的逐元素交互操作表示异构图中环状RNA-疾病关联信息,最后利用多层感知机学习交互的特征信息预测潜在的环状RNA-疾病关联得分。在不同数据集上的五折交叉验证实验表明,GATCL2CD预测性能优于其它的对比方法,案例分析研究进一步证明了GATCL2CD是预测环状RNA与疾病关系的有效工具。
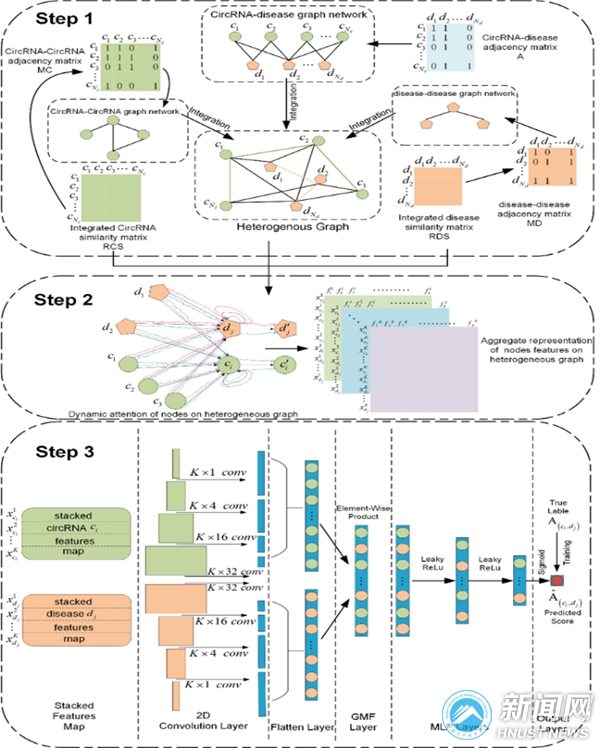
▲GATCL2CD模型总体框架
环状RNA与癌症等人类复杂疾病密切相关,是一种潜在的疾病诊断标志物和药物治疗靶点。由于已知的环状 RNA 与疾病的关联数据非常稀疏,大量假阴性数据的存在使模型的预测性能受到限制。该研究基于异构图注意力网络的特征卷积学习模型对未知的环状RNA -疾病关联进行预测,通过聚合局部网络中邻居节点的特征信息,有效提高了预测准确性,该研究能够为湿实验提供有效指导,对于揭示环状RNA和人类疾病之间的潜在关系以及疾病辅助诊断治疗具有重要意义。
该工作获得国家自然科学基金项目(61902125, 62002111),湖南省教育厅重点项目(22A0350,22A0101)的资助。
论文链接:https://ieeexplore.ieee.org/document/10079096
(一审:李海屿 二审:王 虎 三审:李友桥)